2022年4月29日,js345线路检测王宽诚讲席教授邵志峰与长聘副教授Daniel Czajkowsky领导的科研团队在国际著名期刊《ACS Nano》上在线发表了题为“Single-Molecule Micromanipulation and Super-Resolution Imaging Resolve Nanodomains Underlying Chromatin Folding in Mitotic Chromosomes”的学术论文,报道了运用超分辨显微成像以及结合单分子显微镜的多模态技术研究哺乳动物细胞中期染色体高级结构的重要研究进展。论文第一作者为系统生物医学研究院博士研究生王佳斌,js345线路检测邵志峰教授、Daniel Czajkowsky博士和系统生物医学研究院孙洁林研究员为论文共同通讯作者。

人类基因组DNA在细胞有丝分裂中如何进行三维折叠以及高度压缩形成众所周知的棒状中期染色体是目前细胞生物学中最古老的研究问题之一。错误的折叠压缩结构会导致细胞周期中遗传物质的不正确传递,从而引发癌症或其它疾病。染色体的发现已经超过了100年,人们已经知道200个碱基对的DNA会缠绕在核心组蛋白从而形成10纳米尺度的“念珠”状的核小体,然而核小体是如何进一步缠绕形成更高级的空间结构,最后成为中期染色体的,一直以来困扰着科学家们。
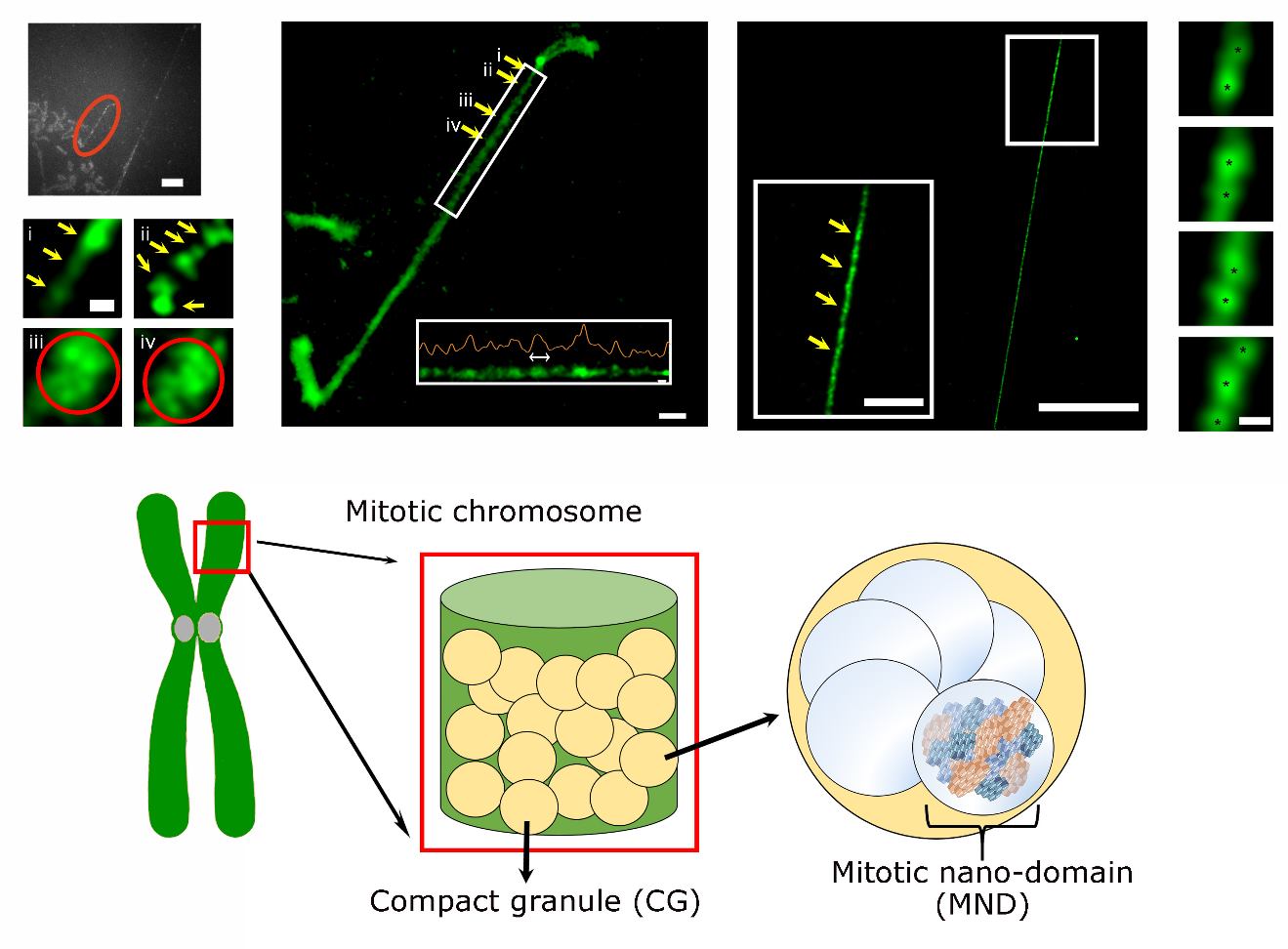
项目团队开发了一种新的多模态技术,最终破解了染色体的物理结构。通过多种超分辨显微成像的方法,他们发现了一种约125纳米的致密颗粒状结构(Compact Granules, CGs), 表明了DNA压缩成为CGs是染色体折叠过程中普遍的形式。进一步解析更精细的结构特征,他们建立了一种新的方法,能够通过玻璃微吸管对单条染色体进行拉伸解构,并且结合超分辨成像技术进行观察。通过这种方法,他们发现CGs展开成为单个的90nm的纳米结构域,取名为有丝分裂纳米结构域(Mitotic nanodomains, MNDs)。令人意外的是,这种MNDs结构能够对抗很大的机械力,暗示它们具有很高的稳定性。因此,他们发现在中期染色体上存在两个层次的高级结构:基本结构MNDs和MNDs组装而成的CGs。
该团队还进一步确定了MNDs的DNA含量约为80kb,并且能够使用MNDs的最密堆积来解释整个基因组的DNA含量和染色体体积。同时,MNDs的DNA含量也与现在流行的染色体结构模型所预测的环状结构DNA含量一致,而此前该模型一直缺乏直接的实验验证。因此,本研究可以与已有模型整合和解释已有的实验数据,为结构生物学中关键的问题之一提供了一种统一的描述。
该项研究得到科技部重点研发计划、国家自然科学基金、js345线路检测和香港王宽诚教育基金会等项目资助。
Paper link: https://doi.org/10.1021/acsnano.2c01025
